![]()
![]()
การแสดงออกของยีนเพื่อสร้างโปรตีนแต่ละชนิดจะเกิดขึ้นในเวลาและปริมาณที่แตกต่างกัน ทั้งนี้ขึ้นกับบทบาทและหน้าที่ของโปรตีนชนิดนั้นๆ โปรตีนที่มีความสำคัญต่อการดำรงชีวิตของเซลล์จะมีการผลิตอยู่ตลอดเวลา ซึ่งการแสดงออกของยีนเพื่อสร้างหรือผลิตโปรตีนเหล่านี้จะเรียกว่า "constitutive gene expression" เช่น การแสดงออกของยีนเพื่อผลิตเอนไซม์ในวิถีไกลโคไลซิส และวัฎจักรเครปส์ และเรียกยีนของโปรตีนเหล่านี้ว่า "house keeping gene"
ส่วนโปรตีนอีกจำพวกหนึ่งจะถูกสังเคราะห์ขึ้นก็ต่อเมื่อเซลล์มีความจำเป็นต้องใช้ หรือถูกเหนี่ยวนำให้สร้างขึ้นในบางสภาวะเท่านั้น เรียกโปรตีนเหล่านี้ว่า "inducible protein" เช่น กลุ่มเอนไซม์ที่ใช้ในการซ่อมแซม DNA จะถูกเหนี่ยวนำให้สร้างขึ้นก็ต่อเมื่อ DNA ถูกทำลาย ในทำนองกลับกัน จะมีโปรตีนอีกจำพวกหนึ่งถูกสร้างน้อยลงเมื่อเซลล์ไม่จำเป็นต้องใช้ เช่น กลุ่มเอนไซม์ที่ใช้ในการสังเคราะห์กรดอะมิโนทริปโตเฟนในแบคทีเรีย จะถูกลดการสร้างลงเมื่อเซลล์มีปริมาณทริปโตเฟนเพียงพอ เรียกโปรตีนเหล่านี้ว่า "repressible protein"
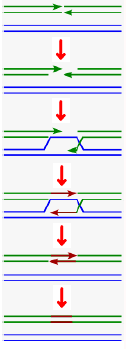
กระบวนการต่างๆ ที่เกิดขึ้นระหว่างการแสดงออกของยีน จะมีผลโดยตรงต่อปริมาณโปรตีนที่เป็นผลิตภัณฑ์ขั้นสุดท้ายทั้งสิ้น การควบคุมการแสดงออกของยีน จะเกี่ยวข้องกับกระบวนการกระตุ้นหรือยับยั้งการสังเคราะห์ RNA และโปรตีนของยีนนั้นๆ (หากยีนนั้นมีไว้สำหรับสร้างโปรตีน) โดยอาศัยการเหนี่ยวนำให้เอนไซม์ทำงานด้วยตัวเหนี่ยวนำ (inducer) หรือยับยั้งการทำงานของเอนไซม์ด้วยตัวกดดัน (repressor) ซึ่งสามารถควบคุมได้ในขั้นตอนการลอกแบบ (transcription) และการแปลรหัส (translation) (ภาพที่ 3.25)
ภาพที่ 3.25 การควบคุมการแสดงออกของยีนในขั้นต่างๆ
![]()
คำถาม : |
การตัดแต่ง
mRNA ถือเป็นการควบคุมการแสดงออกของยีนหรือไม่ ลองหาเหตุผลมาอธิบายว่าทำไมต้องมีการตัดแต่ง
mRNA ในแต่ละขั้น |

โปรคาริโอตมีพันธุกรรมที่ไม่ซับซ้อน กระบวนการสังเคราะห์ RNA และโปรตีนเกิดขึ้นในเวลาที่ไล่เลี่ยกัน ดังนั้นการควบคุมการผลิตโปรตีนในเซลล์โปรคาริโอตจึงเป็นระบบ 'เปิด-ปิด' ทั้งนี้เพื่อให้สามารถเปลี่ยนกลับไป-มาได้ง่ายในเวลาสั้นๆ ดังนั้นการควบคุมการแสดงออกของยีนในโปรคาริโอตจะเกิดในขั้นคัดลอก (transcription)
ในโปรคาริโอต ยีนของโปรคาริโอตสำหรับสร้างโปรตีนที่เกี่ยวข้องในวิถีเมทาบอลิซึมเดียวกัน ส่วนใหญ่จะอยู่ติดกันเป็นชุดและอยู่ภายใต้ตัวควบคุมชุดเดียวกัน รวมเรียกว่าโอเปอรอน (operon) ซึ่ง แบ่งเป็นยีนควบคุม (regulator gene) ยีนส่งเสริม (promoter gene) ยีนดำเนินการ (operator gene) และยีนโครงสร้าง (structural gene) สำหรับสร้างโปรตีนหรือเอนไซม์ (ภาพที่ 3.26)
-
ยีนควบคุม (regulator gene) มีหน้าที่ควบคุมยีนดำเนินการ โดยสร้างตัวกดดัน ซึ่งสามารถจับกับยีนดำเนินการ ทำให้เอนไซม์อาร์เอนเอโพลีเมอเรสไม่สามารถสังเคราะห์ RNAได้
-
ยีนส่งเสริม (promoter gene) เป็นตำแหน่งให้เอนไซม์อาร์เอนเอโพลีเมอเรสจับ และเคลื่อนที่ไปยังยีนโครงสร้างเพื่อสังเคราะห์ RNA แต่ถ้ามีตัวกดดันจับอยู่ที่ยีนดำเนินการ เอนไซม์อาร์เอนเอโพลีเมอเรสก็จะจับกับยีนส่งเสริมไม่ได้ เป็นการยับยั้งการสังเคราะห์ RNA
-
ยีนดำเนินการ (operator gene) อยู่ถัดจากยีนส่งเสริมและอยู่ก่อนหน้ายีนโครงสร้าง เป็นตำแหน่งจับของตัวกดดันในการควบคุมการสังเคราะห์ RNA
-
ยีนโครงสร้าง (structural gene) เป็นยีนหลักที่มีข้อมูลทางพันธุกรรมซึ่งจะสร้างโปรตีนโดยตรง การแสดงออกของยีนโครงสร้างจะผ่านทางการควบคุมการทำงานของยีนต่างๆ ที่กล่าวมาข้างต้น

ภาพที่ 3.26 โครงสร้างของโอเปอรอน
|
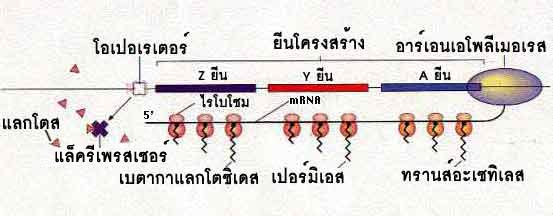
ตัวอย่างของโอเปอรอนของการสร้างเอนไซม์ที่ใช้อธิบายทั่วไปคือ แล็คโอเปอรอน (lac operon) และ ทริปโอเปอรอน (trp operon)การทำงานของแล็คโอเปอรอนอาศัยตัวเหนี่ยวนำ ขณะที่ทริปโอเปอรอนทำงานโดยอาศัยตัวกดดัน แล็คโอเปอรอน (lac operon) เป็นโอเปอรอนที่เกี่ยวข้องกับการสังเคราะห์เอนไซม์ที่ใช้สำหรับเมทาบอลิซึมของน้ำตาลแล็คโตส ซึ่งยีนโครงสร้างที่เป็นองค์ประกอบของแล็คโอเปอรอน ได้แก่ - beta-galcatosidase (Z) gene สำหรับสังเคราะห์เอนไซม์ beta-galcatosidase เพื่อทำหน้าที่ย่อยสลายแล็คโตส ให้เป็นกาแล็คโตส และกลูโคส - galactosidase permease (Y) gene สำหรับสังเคราะห์เอนไซม์ galactosidase permease ซึ่งทำหน้าที่เกี่ยวข้องกับการนำแล็คโตสเข้าสู่เซลล์ - thiogalactosidase tranacetylase (A) gene สำหรับสร้างเอนไซม์ thiogalactosidase tranacetylase ซึ่งทำหน้าที่เติมหมู่อะซิทิลให้กับ galactoside ที่ไม่ถูกย่อยและนำส่งออกนอกเซลล์ ส่วนของดีเอนเอที่ทำหน้าที่ควบคุมแล็คโอเปอรอน จะอยู่ตอนต้นของยีนโครงสร้าง โดยประกอบด้วยโปรโมเตอร์ (P) ซึ่งเป็นบริเวณที่เอนไซม์ RNA polymerase เข้าจับเพื่อเริ่มต้นการถอดรหัส และโอเปอเรเตอร์ (O) ซึ่งเป็นบริเวณที่โปรตีนกดดัน (repressor) เข้าจับ โดยในสภาวะที่เซลล์ไม่มีแล็คโตส แบคทีเรีย E. coli ไม่จำเป็นต้องใช้กลุ่มเอนไซม์ในแล็คโอเปอรอน ดังนั้นการสังเคราะห์ mRNA สำหรับเอนไซม์ในแล็คโอเปอรอน จะถูกยับยั้งด้วยโปรตีนแล็ครีเพรสเซอร์ (lac repressor) ซึ่งสังเคราะห์มาจากยีนควบคุม (I) โดยแล็ครีเพรสเซอร์จะเข้าจับกับโอเปอเรเตอร์ และยับยั้งการทำงานของเอนไซม์ RNA polymerase ในภาวะที่มีน้ำตาลแล็คโตส จะมีการกระตุ้นแล็กโอเปอรอนให้มีการสังเคราะห์กลุ่มเอนไซม์ที่จำเป็นในการใช้แล็กโตส โดยโมเลกุลของแล็กโตสจะเข้าจับกับแล็ครีเพรสเซอร์ ทำให้โมเลกุลของแล็ครีเพรสเซอร์เกิดการเปลี่ยนแปลงโครงสร้าง ไม่สามารถจับกับโอเปอเรเตอร์ได้อีกต่อไป ส่งผลให้เซลล์สามารถสังเคราะห์กลุ่มเอนไซม์ที่จำเป็นในการใช้แล็กโตสได้ ซึ่งในกรณีนี้จะเรียกแล็กโตสว่าเป็นตัวเหนี่ยวนำ (inducer) (ภาพที่ 3.27)
|
||||||||||
ภาพที่ 3.27 โครงสร้างของแล็คโอเปอรอน |
||||||||||
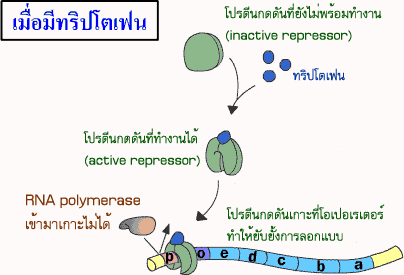
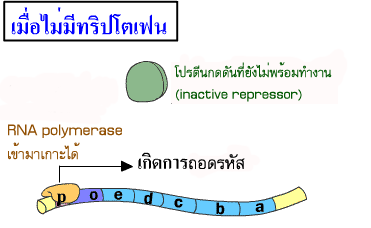
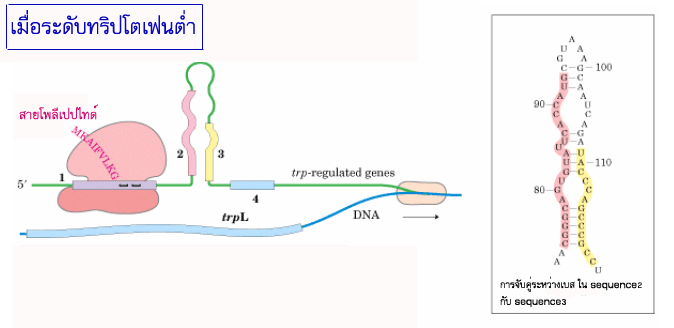
ทริปโอเปอรอน (trp operon) เป็นโอเปอรอนที่เกี่ยวข้องกับการสังเคราะห์กลุ่มของเอนไซม์ที่ใช้ในการสังเคราะห์กรดอะมิโนทริปโตเฟน (tryprophan) โดยองค์ประกอบหลักของทริปโอเปอรอน จะคล้ายกับ lac operon แต่มี attenuator ทำหน้าที่ควบคุมเพิ่มขึ้นอีกส่วนหนึ่ง
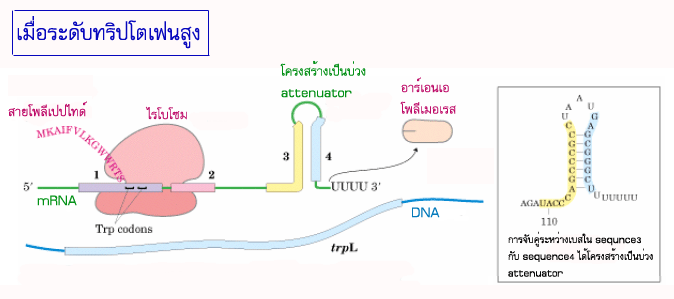
การสังเคราะห์เอนไซม์ในทริปโอเปอรอนจะดำเนินไปจนกระทั่งระดับทริปโตเฟนในเซลล์สูงเพียงพอต่อความต้องการของเซลล์ จากนั้นจะมีกลไกในการทำให้อัตราการสังเคราะห์เอนไซม์ในทริปโอเปอรอนค่อยๆ ลดลงโดยระบบที่เรียกว่า attenuation ซึ่งเป็นการควบคุมการลอกแบบ โดยอาศัยขั้นตอนของการแปลรหัสมาเกี่ยวข้อง โดยจะมีการควบคุมการลอกแบบหลังจากที่การถอดรหัสได้เริ่มต้นไปแล้ว การควบคุมแบบนี้ เกิดขึ้นเฉพาะในเซลล์โปรคาริโอตเท่านั้น เนื่องจากเซลล์โปรคาริโอตไม่มีเยื่อหุ้มนิวเคลียส ทำให้การสังเคราะห์โปรตีนเกิดขึ้นในขณะที่การสังเคราะห์ mRNA กำลังดำเนินอยู่ ทริปโอเปอรอนประกอบด้วยส่วนของ DNA ประมาณ 162 นิวคลีโอไทด์ ที่ตำแหน่งก่อนหน้ายีนทางด้าน 5' ที่เรียกว่า leader region ซึ่ง leader region นี้จะมีส่วนของนิวคลีโอไทด์ 4 บริเวณ ที่มีเบส G และ C มากเป็นพิเศษ โดยเรียกชื่อเป็น sequence1 sequence2 sequence3 และ sequence4 เรียงลำดับจาก 5' ไปยัง 3' โดยเมื่อบริเวณดังกล่าวถูกถอดรหัสไปเป็น mRNA ลำดับเบสของนิวคลีโอไทด์บน mRNA ในบริเวณที่อยู่ติดกันสามารถจับกันได้ ซึ่งถ้า sequence3 และ sequence4 สามารถจับกันได้ จะเกิดเป็นโครงสร้างคล้ายบ่วงที่เรียกว่า attenuator บนสาย mRNA ซึ่งจะมีผลทำให้ RNA polymerase ที่กำลังทำหน้าที่ลอกแบบบนสาย DNA หลุดออกไป เป็นผลให้การสังเคราะห์ mRNA ถูกชะลอลง (ภาพที่ 3.28) ซึ่งการจับกันเป็นบ่วงของ attenuator ขึ้นกับการแปลรหัสที่บริเวณ sequence1 ของ leader region บนสาย mRNA
|

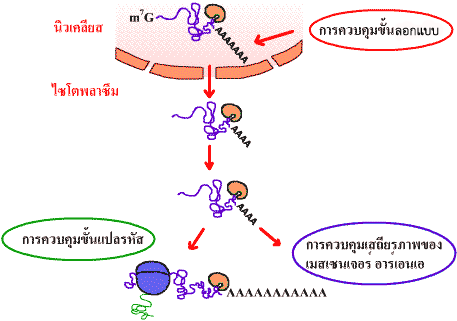
การควบคุมการแสดงออกของยีนในยูคาริโอตจะซับซ้อนกว่าในโปรคาริโอตมาก เนื่องจากจีโนมของยูคาริโอตมียีนมากกว่าในโปรคาริโอตมาก ยีนทั้งหมดนี้ไม่ได้ทำงานพร้อมกัน ซึ่งยีนแต่ละยีนนั้นจะมีการแสดงออกแตกต่างกันตามเวลาและสภาวะของเซลล์ นอกจากนี้การที่เซลล์ยูคาริโอตมีเยื่อหุ้มนิวเคลียส จึงทำให้กระบวนการถอดรหัสเกิดในนิวเคลียส และกระบวนการแปลรหัสเกิดในไซโตพลาสซึมแยกจากกันเด็ดขาด มิได้เกิดต่อเนื่องกันอย่างในโปรคาริโอต
ลักษณะที่แตกต่างระหว่างพันธุกรรมของยูคาริโอต และโปรคาริโอตที่สำคัญอีกประการหนึ่ง คือ ยีนของยูคาริโอตจะอยู่แยกเป็นส่วนๆ ที่เรียกว่าเอกซอน (exon) ซึ่งต้องมีกระบวนการตัดอินทรอน (intron) ออกและเชื่อมเอกซอนเข้าด้วยกัน ด้วยกลไกพื้นฐานที่แตกต่างกันมากระหว่างยูคาริโอต และโปรคาริโอต จึงทำให้การควบคุมการแสดงออกของยีนในยูคาริโอตมีความยุ่งยาก และแตกต่างจากโปรคาริโอต ซึ่งในยูคาริโอตมีการควบคุมการแสดงออกของยีน ทั้งในขั้นถอดรหัสและแปลรหัส
การควบคุมในขั้นคัดลอก (transcription)
การควบคุมการแสดงออกของยีนในยูคาริโอตในขั้นการคัดลอก ได้แก่ การเติมหมู่เมทิล เข้าที่เบสไซโตซีน (cytosine, C) ของดีเอนเอ โดยเอนไซม์ DNA metyltransferase (DNA MTase) กระบวนการเติมหมู่เมทิลเข้าที่เบสของดีเอนเอนี้เรียกว่า DNA metylation บริเวณที่มีเบส CG จำนวนมาก และอยู่ตอนต้นของยีนโครงสร้าง มักจะถูกเติมหมู่เมทิล (ภาพที่ 3.29) ซึ่งจะมีส่วนเกี่ยวข้องกับกระบวนการสังเคราะห์ RNA กล่าวคือ หากเบสไซโตซีนในบริเวณดังกล่าวของยีนใดถูกเติมหมู่เมทิล ยีนนั้นจะไม่ทำงานหรือไม่แสดงออก (ภาพที่ 3.30)
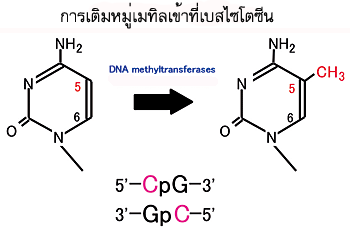
ภาพที่ 3.29 การเติมหมู่เมทิลเข้าที่เบสไซโตซีน
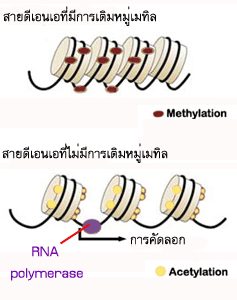
ภาพที่ 3.30 ดีเอนเอที่มีหมู่เมทิล (บน) และดีเอนเอที่ไม่มีหมู่เมทิล (ล่าง)
ความสัมพันธ์ของการเติมหมู่เมทิลของเบสไซโทซีนกับการควบคุมการแสดงออกของยีน ได้รับการพิสูจน์จากการเพาะเลี้ยงเซลล์มะเร็งของต่อมใต้สมองของหนูพบว่า ไซโทซีนในยีนของเซลล์ที่สามารถสร้างฮอร์โมนโพรแลกทิน (prolactin) ไม่มีหมู่เมทิล ในขณะที่ ไซโทซีนจำนวนมากในยีนของเซลล์ที่ไม่สามารถสร้างฮอร์โมนโพรแลกทินมีหมู่เมทิล
กระบวนการเติมหมู่เมทิลให้กับเบสไซโตซีนในสายดีเอนเอนี้ นอกจะทำให้ยีนนั้นไม่แสดงออกแล้ว ยังพบว่ามีบทบาทสำคัญต่อ gene imprinting ซึ่งเป็นลักษณะที่เกิดขึ้นในกระบวนการสร้างเซลล์สืบพันธุ์ โดยโครโมโซม X ตัวที่ไม่ทำงาน (inactive X) จะมีการเติมหมู่เมทิล โดยเชื่อกันว่ากระบวนการนี้เป็นกระบวนการที่จำเป็นในการเจริญเติบโต (development) ของตัวอ่อน
การควบคุมในขั้นแปลรหัส (translation)
การควบคุมการแสดงออกของยีนในระดับการแปลรหัสเกิดขึ้นได้หลายขั้นตอน ขึ้นอยู่กับ 'สัญญาณ' ที่เป็นตัวเริ่มกระบวนการ เช่น เซลล์ไข่เมื่อยังไม่ได้รับการผสมพันธุ์จะมี mRNA อยู่เป็นจำนวนมาก แต่ mRNA เหล่านี้ไม่ถูกนำไปสังเคราะห์เป็นโปรตีน แต่เพียงไม่กี่นาทีหลังการผสมพันธุ์ จะมีการสังเคราะห์โปรตีนจำนวนมากจาก mRNA เหล่านี้ การที่ mRNA เหล่านี้ไม่ถูกนำไปสังเคราะห์เป็นโปรตีนก่อนการผสมพันธุ์นั้น เนื่องจาก mRNA เหล่านั้นจับกับโปรตีนบางชนิด ซึ่งปกปิดหรือซ่อน mRNA เหล่านี้ไว้
กลไกการควบคุมการแสดงออกของยีน อาจใช้ตัวกดดันคือโปรตีนรีเพรสเซอร์ (repressor protein) โดยเมื่อจับกับ mRNA แล้วจะยับยั้งการแปลรหัสได้ ตัวอย่างเช่น การยับยั้งการแปลรหัสของเฟอริติน (ferritin) โดย iron repressive mRNA binding protein ซึ่งจะตอบสนองต่อเหล็ก โดยเมื่อมีเหล็กปริมาณน้อย โปรตีนนี้จะสามารถยับยั้งการแปลรหัสได้
การแสดงออกของยีนในยูคาริโอต นอกจากจะถูกควบคุมโดยโปรตีนแล้ว ในปัจจุบันพบว่า มี RNA บางชนิดสามารถควบคุมการแสดงออกของยีนได้เช่นกัน เช่น siRNA, shRNA และ miRNA โดย RNA เหล่านี้จะทำให้ mRNA ไม่ถูกแปลรหัสออกมาเป็นโปรตีน
siRNA (small interference RNA) เกิดจาก RNA เกลียวคู่ (dsRNA, double strand RNA) ที่มีความยาวมากกว่า 30 นิวคลีโอไทด์ ถูกตัดด้วย dicer ทำให้ได้ siRNA ซึ่งมี 21-23 คู่นิวคลีโอไทด์ และตรงบริเวณปลาย 3' ของ RNA แต่ละสายจะมีมีเบส 2 ตัวยื่นออกมา siRNA จะมีความจำเพาะกับ mRNA เป้าหมาย สามารถทำให้ยีนบน mRNA เป้าหมายไม่สามารถแสดงออกได้ โดย siRNA จะจับกับโปรตีนได้เป็น RNA-Protein complex ที่มีชื่อว่า RISC (RNA induce silencing complex) เมื่อมีการจับตัวกันเป็นโครงสร้างที่ซับซ้อนแบบนี้แล้ว siRNA จะคลายเกลียว ทำให้สามารถจับกับ mRNA เป้าหมายได้ โดยเป็นการจับกันระหว่างเบสในสาย mRNA และ siRNA ที่เป็นคู่สมกัน หลังจากนั้นสายของ mRNA เป้าหมายนี้ก็จะถูกตัดโดย RISC ซึ่งมีเอนไซม์นิวคลีเอส (nuclease) เป็นตัวทำหน้าที่ในการตัด (ภาพที่ 3.31)
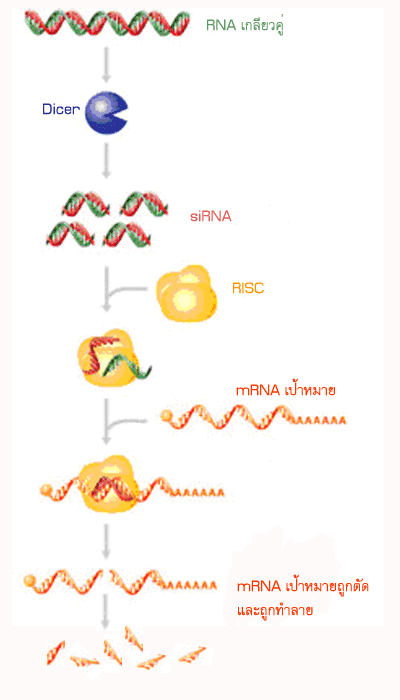
ภาพที่ 3.31กลไกควบคุมการแสดงออกของยีนโดย siRNA (small interfering RNA)
| การยับยั้งการแสดงออกของยีนโดย RNA เกลียวคู่นี้ ถูกค้นพบโดยนักวิทยาศาสตร์ชาวอเมริกัน 2 ท่าน คือ ไฟร์ (Andrew Z. Fire) และเมลโล (Craig C. Mello) ซึ่งทำให้พวกเขาได้รับรางวัลโนเบลทางการแพทย์ในปี 2006 (พ.ศ.2549) |
|
shRNA (short hairpin RNA) มีโครงสร้างเป็น stem-loop มีประมาณ 25-29 นิวคลีโอไทด์ ตรงปลาย 3 จะมีเบสยูราซิล 2 ตัวยื่นออกมา (3 overhang) นิวคลีโอไทด์ที่อยู่ตรง loop นี้ จะถูกตัดออกภายหลัง ทำให้เหลือประมาณ 21 นิวคลีโอไทด์ซึ่งมีโครงสร้างเหมือนกับ siRNA ดังนั้นมันจึงมีความสามารถในการยับยั้งการแสดงออกของยีนในยูคาริโอตได้เช่นเดียวกับ siRNA (ภาพที่ 3.32)
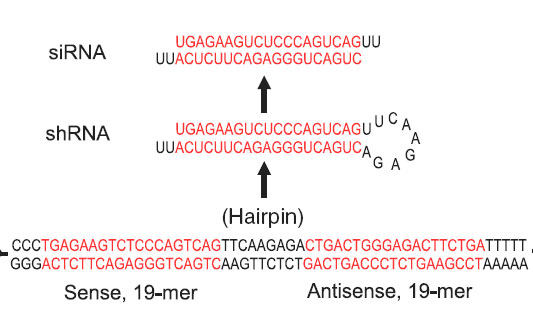
ภาพที่ 3.32 ตัวอย่าง small hairpin RNA (shRNA)
.
miRNA (micro RNA) เป็น RNA สายเดี่ยวเส้นสั้นๆ ที่มี 19 - 23 นิวคลีโอไทด์ ถูกถอดรหัสออกมาเป็นส่วนหนึ่งของสาย RNA ที่มี 100 นิวคลีโอไทด์ หลังจากนั้นจะฟอร์มรูปร่างเป็น hiarpin ภายในนิวเคลียส แล้วถูกส่งออกสู่ไซโตพลาสซึม หลังจากนั้นมันจะถูกตัดด้วย dicer ได้เป็น RNA สายเดี่ยวหรือ miRNA นั่นเอง ซึ่ง miRNA มีบทบาทในการควบคุมการแสดงออกของยีนในยูคาริโอตเช่นเดียวกับ siRNA และ shRNA แต่จะมีกลไกไกที่แตกต่างกันคือ miRNA จะไม่ทำลายสายของ mRNA เป้าหมาย แต่มันจะจับกับ mRNA เป้าหมาย ทำให้การแปลรหัสถูกยับยั้ง ดังนั้นการสร้างโปรตีนจึงไม่เกิดขึ้น (ภาพที่ 3.33)
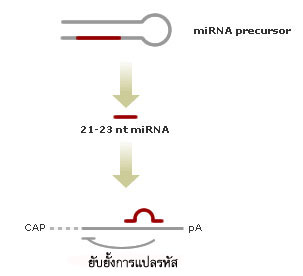
ภาพที่ 3.33 กลไกการควบคุมการแสดงออกของยีนโดย miRNA (micro RNA)
นอกเหนือจากการค้นพบ small interference RNA ตัวเล็กๆ หรือ siRNA short hairpin RNA (shRNA) และ micro RNA (miRNA) ดังกล่าวแล้ว ในระยะหลังยังพบอาร์เอ็นเอทั้งเล็กและใหญ่ที่ไม่ได้ทำหน้าที่เกี่ยวข้องกับการสร้างโปรตีนเลย คือไม่ใช่ mRNA tRNA หรือ mRNA แต่ทำหน้าที่ที่เกี่ยวกับการควบคุมการทำงานของยีนและการทำงานของ mRNA ที่น่าทึ่งกว่านี้คือ อาร์เอ็นเอเหล่านี้มีความหลากหลายและมีปริมาณในส่วนต่างๆ ของเซลล์มากกว่าที่นักวิทยาศาสตร์เคยคิด ตัวอย่างเช่น scnRNA tasiRNA natsiRNA และ piRNA ต่างทำหน้าที่เกี่ยวกับการเจริญเติบโตและการพัฒนาของเซลล์ XIST RNA ทำงานโดยปิดการทำงานของโครโมโซม PINC RNA เกี่ยวกับการตั้งครรภ์ ขณะนี้มีการคาดคะเนว่ายีนที่ทำหน้าที่เกี่ยวกับการสร้างโปรตีนอาจจะมีจำนวนน้อยกว่ายีนที่ถูกคัดลอกไปเป็นอาร์เอ็นเอที่ไม่เกี่ยวกับการสร้างโปรตีนโดยตรง เช่น ในคนอาจมียีนสำหรับอาร์เอ็นเออื่นๆ ที่ไม่เกี่ยวกับการสร้างโปรตีนถึงประมาณ 37,000 ชนิด ซึ่งเป็นจำนวนมากกว่ายีนสำหรับสร้างโปรตีนซึ่งมีเพียงประมาณ 21,000 ชนิด เท่านั้น

ยีนมิวเทชันหรือการกลายของยีน คือการที่ยีนมีโครงสร้างผิดปกติไปจึงทำหน้าที่ต่างไปจากเดิม การกลายของยีนมีหลายประเภท หากแบ่งตามปัจจัยที่ทำให้เกิดได้แก่ รังสี (ภาพที่ 3.34) สารเคมี (ภาพที่ 3.35) รวมไปถึงจุลินทรีย์ที่เป็นปรสิต เช่นไวรัส
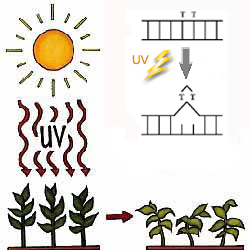
ภาพที่ 3.34 การกลายของยีนเนื่องจากการสัมผัสรังสีอัลตร้าไวโอเล็ต
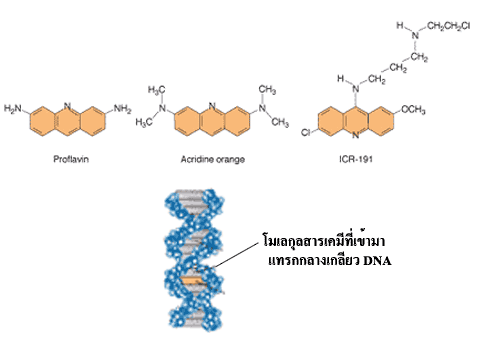
ภาพที่ 3.35 การกลายของยีนเนื่องจากสารเคมีเข้าไปแทรกอยู่ในโมเลกุล DNA
หากแบ่งการกลายของยีนตามลักษณะการเกิดได้แก่ การกลายเนื่องจากการแทนที่เบส (ภาพที่ 3.36) และการขาดหรือเกินของเบส (ภาพที่ 3.37)
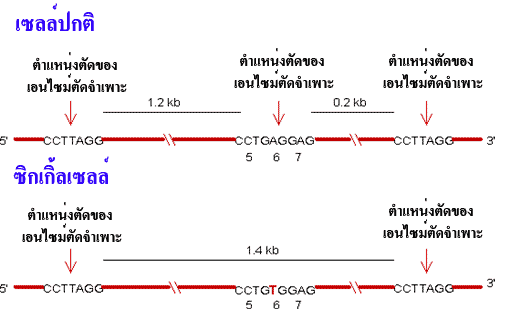
ภาพที่ 3.36 การกลายของยีนเนื่องจากการแทนที่เบส
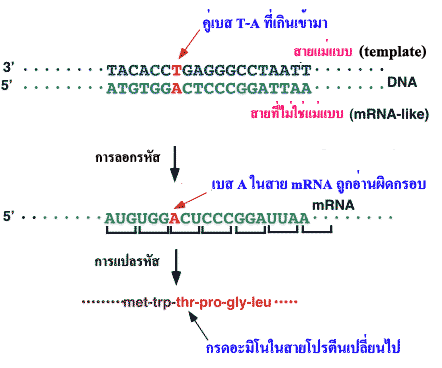
ภาพที่ 3.37 การกลายของยีนเนื่องจากการขาดหรือเกินของเบส
![]()
คำถาม : |
ลองยกตัวอย่างการกลายของยีนประเภทต่างๆ
เพิ่มเติม พร้อมทั้งอธิบายผลที่เกิดขึ้น และหากการกลายลักษณะต่างๆ
ที่กล่าวมาเกิดกับ DNA ส่วนที่ไม่ใช่ยีนจะมีผลต่างกับการกลายที่เกิดกับยีนอย่างไร |
การซ่อมแซม DNA เป็นกระบวนการที่เซลล์ใช้รักษาสภาพไว้ให้คงเดิมหลังจากเกิดการกลาย (มิวเตชัน) ขึ้น ซึ่งการซ่อมแซมนี้มีหลายประเภทขึ้นกับความผิดปกติที่เกิดขึ้น สามารถแบ่งมิวเทชันเป็นประเภทใหญ่ๆได้ 4 ประเภทคือ
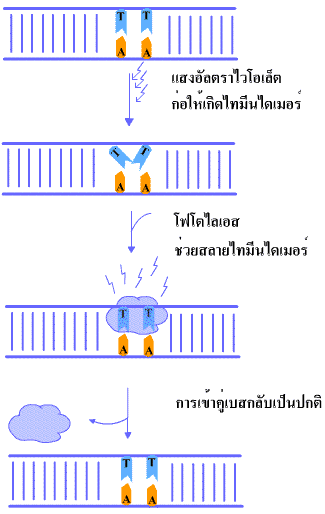
โฟโตรีแอคติเวชัน (photoreactivation repair) เป็นการบวนการซ่อมแซมดีเอนเอโดยการใช้แสงในช่วงคลื่นที่มองเห็น (visible light) สลายพันธะไพริมีดีนไดเมอร์ที่เกิดขึ้นในสาย DNA โดยใช้เอนไซม์โฟโตไลเอส (ภาพที่ 3.38) กระบวนการดังกล่าวพบเฉพาะในแบคทีเรีย
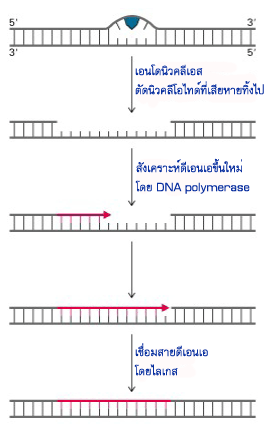
เอ็กซ์ซิชันรีแพร์ (excision repair) เป็นกระบวนการซ่อมแซมโดยมีการตัดเอาเบสหรือนิวคลีโอไทด์ที่เสียหายออกก่อน แล้วจึงเติมเบสหรือนิวคลีโอไทด์ที่ถูกต้องเข้าไปใหม่ (ภาพที่ 3.39)
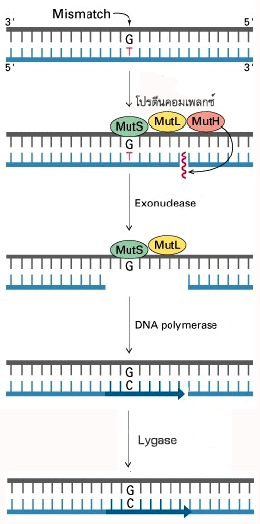
มิสแมทช์รีแพร์ (mismatch repair) เป็นกระบวนการกำจัดเบสที่เข้าคู่ผิดในขณะเกิดการลอกแบบทิ้งไป แล้วนำเบสที่ถูกต้องมาเข้าคู่แทน (ภาพที่ 3.40)
รีคอมบิเนชันรีแพร์ (recombination repair) เป็นกระบวนการซ่อมแซม DNA ที่เข้าคู่ผิดภายหลังการลอกแบบ โดยอาศัย DNA ในโฮโมโลกัสโครโมโซมที่ถูกต้องเป็นแม่แบบ (ภาพที่ 3.41)
ภาพที่ 3.38 การซ่อมแซมแบบโฟโตรีแอคติเวชัน (photoreactivation repair) |
ภาพที่ 3.39 การซ่อมแซมโดยการตัดออกหรือเอ็กซ์ซิชัน (excision repair) |
ภาพที่ 3.40 การซ่อมแซมส่วนที่จับผิดคู่หรือมิสแมทช์ (mismatch repair) |
ภาพที่ 3.41 การซ่อมแซมโดยการแลกชิ้นส่วนดีเอนเอหรือรีคอมบิเนชัน (recombination repair) |
![]()
คำถาม : |
การซ่อมแซม
DNA แบบใดที่น่าจะเกิดขึ้นบ่อยที่สุดในเซลล์ที่กำลังแบ่งตัว เพราะเหตุใด |
| ภาพยนตร์ที่เกี่ยวข้องกับบทเรียน |
|
ชื่อเรื่อง |
ขนาด |
| lac operon movie | 2,484 KB |
| trp operon movie | 5,463 KB |