การสร้างออร์แกเนลล์เพื่อเตรียมพร้อมสำหรับการแบ่งเซลล์ ระยะนี้แบ่งออกเป็น 3 ระยะย่อยๆ
ได้แก่ ระยะ G1 ระยะ S และระยะ G2 ในทั้ง 3 ระยะนี้เซลล์จะมีการสังเคราะห์โปรตีน
คาร์โบไฮเดรต ไขมัน และอื่นๆ เพื่อใช้สร้างองค์ประกอบที่จำเป็นสำหรับเซลล์ เช่น
เยื่อหุ้มเซลล์ รวมทั้งออร์แกเนลล์อื่นๆ แต่การลอกแบบดีเอ็นเอ (DNA replication) เพื่อเพิ่ม
ปริมาณดีเอ็นเอหรือสารพันธุกรรมนั้นจะเกิดขึ้นในระยะ S เท่านั้น (S ย่อมาจาก
synthesis แปลว่าสังเคราะห์) ในระยะอินเตอร์เฟสนี้จะสามารถสังเกตเห็นขอบเขตของ
นิวเคลียสได้ชัดเจน เนื่องจากเยื่อหุ้มนิวเคลียสยังไม่สลายในไซโทพลาซึม เซนโทรโซมที่เคย
มีหนึ่งอันจะจำลองตัวเองจนมีสองอัน ในเซลล์สัตว์ตรงกลางของเซนโทรโซมจะมีเซนทริโอล
(centriole) 2 อันวางตั้งฉากซึ่งกันและกัน ไมโครทูบูล (microtubule) ซึ่งทำหน้าที่เกี่ยวกับ
การแยกโครโมโซมจะยืดยาวออกมาจากบริเวณเซนโทรโซม ส่วนในเซลล์พืชชั้นสูงจะไม่พบ
โครงสร้างที่มีลักษณะดังกล่าว ในช่วงท้ายของระยะนี้แม้ว่าการลอกแบบดีเอ็นเอจะเสร็จเรียบร้อยแล้ว แต่เราจะไม่สามารถสังเกตเห็นโครโมโซมเป็นแท่งๆ เหมือนเครื่องหมายกากบาท เนื่องจากดีเอ็นเอยังอยู่ในสภาพโครมาทินที่ยังไม่ขดตัวกันแน่น
ในแต่ละรอบของการแบ่งเซลล์แบบไมโทซีสระยะอินเตอร์เฟสจะใช้เวลายาวนานกว่า
90
เปอร์เซ็นต์ของเวลาทั้งหมด และแม้จะเป็นระยะที่ไม่สามารถสังเกตเห็นการเปลี่ยนแปลงภายใต้
กล้องจุลทรรศน์ได้อย่างชัดเจน แต่ก็เป็นระยะที่มีความสำคัญมากเนื่องจากเป็นระยะที่มี
การเพิ่มปริมาณดีเอนเอหรือโครมาทินขึ้นเป็นสองเท่า (แต่จำนวนชุดของสารพันธุกรรม
ยังเท่ากับ 2n เช่นเดิม) เช่น ถ้ากำหนดให้ตัวอักษรภาษาอังกฤษ 1 ตัว แทนโครมาทิน 1 สาย
ในระยะอินเตอร์เฟส สิ่งที่เกิดขึ้นคือ
เปอร์เซ็นต์ของเวลาทั้งหมด และแม้จะเป็นระยะที่ไม่สามารถสังเกตเห็นการเปลี่ยนแปลงภายใต้
กล้องจุลทรรศน์ได้อย่างชัดเจน แต่ก็เป็นระยะที่มีความสำคัญมากเนื่องจากเป็นระยะที่มี
การเพิ่มปริมาณดีเอนเอหรือโครมาทินขึ้นเป็นสองเท่า (แต่จำนวนชุดของสารพันธุกรรม
ยังเท่ากับ 2n เช่นเดิม) เช่น ถ้ากำหนดให้ตัวอักษรภาษาอังกฤษ 1 ตัว แทนโครมาทิน 1 สาย
ในระยะอินเตอร์เฟส สิ่งที่เกิดขึ้นคือ

รูปที่ 1.6 แสดงจำนวนชุดของสารพันธุกรรมในการแบ่งตัวระยะอินเตอร์เฟส
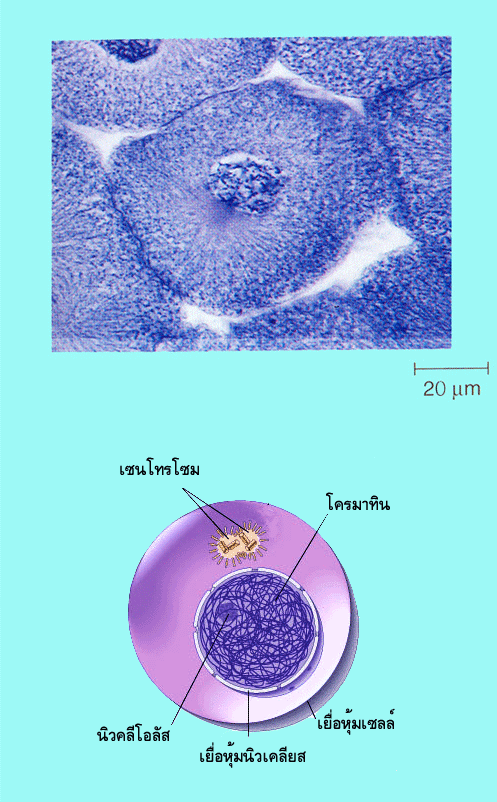
รูปที่ 1.7 การแบ่งเซลล์ในระยะอินเตอร์เฟส
 |
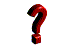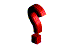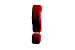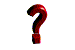 |
: คุณคิดว่าเพราะเหตุใด หรือจะเกิดผลดีอย่างไรกับเซลล์ที่ใช้เวลาเกือบทั้งหมด ในการแบ่งเซลล์แต่ละรอบไปกับระยะอินเตอร์เฟส |
มากที่สุดคือ การลอกแบบดีเอ็นเอ (DNA replication) จึงขอกล่าวในรายละเอียดเพื่อประกอบ
การทำความเข้าใจดังนี้
ดีเอ็นเอให้มากขึ้น เพื่อถ่ายทอดไปสู่ลูกหลานต่อไปในกระบวนการแบ่งเซลล์ แม้ว่าวิธีการ
ดังกล่าวนี้จะมีความแตกต่างกันบ้างในแต่ละสิ่งมีชีวิต แต่สิ่งหนึ่งที่เหมือนกันคือ มีการใช้
ดีเอ็นเอสายเก่าเป็นแม่แบบในการสร้างสายใหม่ทุกครั้ง ดังนั้น DNA ใหม่ที่ได้ 2 สายจึง
ประกอบด้วยโพลีนิวคลีโอไทด์สายเก่า 1 สาย และสายใหม่ 1 สาย เรียกการลอกแบบลักษณะ
นี้ว่าการลอกแบบกึ่งอนุรักษ์ (semiconservative replication) การทดลองทางวิทยาศาสตร์
ที่สำคัญ ที่เป็นเครื่องยืนยันความถูกต้องของการลอกแบบดีเอ็นแบบกึ่งอนุรักษ์ ได้แก่
การทดลองของ Matthew Meselson และ Franklin Stahl
รูปที่ 1.8 การลอกแบบดีเอ็นเอ
การลอกแบบของดีเอ็นเอ มีขั้นตอนต่างๆ ที่สำคัญดังนี้
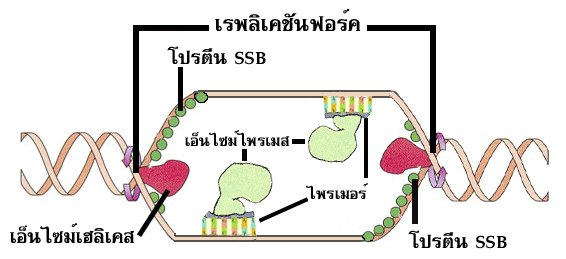
1. มีโปรตีนที่ทำหน้าที่เกี่ยวข้องกับการเริ่มต้นการลอกแบบดีเอ็นเอจำนวนหนึ่ง เข้าจับกับ
สายดีเอ็นเอตรงตำแหน่งจำเพราะที่เรียกว่า จุดเริ่มต้นของการลอกแบบดีเอ็นเอ (origin of
replication) ซึ่งในโปรแคริโอตมีจุดเริ่มต้นของการลอกแบบดีเอ็นเอ 1 จุด ในขณะที่ยูแคริโอต
มีจุดเริ่มต้นของการลอกแบบดีเอ็นเอมากกว่า 1 จุดบนดีเอ็นเอเกลียวคู่ 1 สาย โปรตีนเหล่านี้
มีหน้าที่ในการง้างดีเอ็นเอเกลียวคู่นั้นให้แยกออกจากกันเพื่อให้เอ็นไซม์เฮลิเคส (helicase)
เข้าจับกับสายดีเอ็นเอและสลายพันธะไฮโดรเจนระหว่างคู่เบสบนสายดีเอ็นเอในบริเวณนั้น
ต่อไปการแยกออกจากกันของดีเอ็นเอเกลียวคู่จะทำให้เกิดตำแหน่งที่มีลักษณะคล้ายตัว Y
ขึ้น 2 จุด (ด้านซ้ายและด้านขวาของจุดเริ่มต้นของการลอกแบบดีเอ็นเอ) เรียกจุดที่มีลักษณะ
ดังกล่าวนี้ว่า เรพลิเคชันฟอร์ค (replication fork)


รูปที่ 1.9 ขั้นตอนของการลอกแบบของดีเอ็นเอ
ที่แยกออกจากกัน ทั้งนี้เพื่อป้องกันไม่ให้ดีเอ็นเอสายเดี่ยวทั้งสองสายนั้นกลับมาพันเกลียวกัน
อีกครั้งก่อนที่จะเกิดการลอกแบบดีเอ็นเอ

รูปที่ 1.10 การจับของโปรตีน SSB ที่เรพลิเคชันฟอร์ด
อาร์เอ็นเอไพรเมอร์ (RNA primer) โดยการทำงานของเอ็นไซม์อาร์เอ็นเอไพรเมส
(RNA primase) อาร์เอ็นเอสายสั้นๆ ที่เกิดขึ้นนี้จะทำหน้าที่เป็นจุดเริ่มต้นการทำงานของ
เอ็นไซม์ดีเอ็นเอโพลีเมอเรส ซึ่งเป็นเอ็นไซม์ที่ทำหน้าที่ในการนำดีออกซีไรโบนิวคลีโอไทด์
เดี่ยว เข้ามาต่อเพื่อให้เกิดเป็นดีเอ็นเอสายยาว การทำงานที่ต้องอาศัยอาร์เอ็นเอไพรเมอร์นี้
จึงเป็นข้อแตกต่างอย่างหนึ่งระหว่างเอ็นไซม์ดีเอ็นเอโพลีเมอเรส กับเอ็นไซม์อาร์เอ็นเอ
โพลีเมอเรส ซึ่งทำหน้าที่ในการคัดลอกดีเอ็นเอเพื่อสังเคราะห์เป็็นอาร์เอ็นเอ
รูปที่ 1.11 ขั้นตอนการสร้าง RNA primer
และเริ่มการนำดีออกซีไรโบนิวคลีโอไทด์เดี่ยว (deoxyribonucleotide) เข้ามาต่อสาย
ในทิศทาง 5'-3' โดยเชื่อมเบสที่คู่กันเข้าด้วยกันด้วยพันธะไฮโดรเจน และเชื่อมหมู่ฟอสเฟต
ของแต่ละนิวคลีโอไทด์ ให้เป็นพันธะฟอสโฟไดเอสเทอร์ เมื่อจุดคลายเกลียวเลื่อนขึ้นไป
ก็จะได้ดีเอ็นเอสายใหม่ที่ยาวขึ้นเรื่อยๆ ตามทิศทางการเคลื่อนที่ของเรพลิเคชันฟอร์ค
เรียกดีเอ็นเอสายนี้ว่า สายใหม่ที่นำหน้าการลอกแบบ (leading strand)
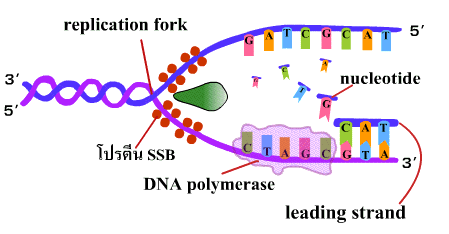
รูปที่ 1.12 การสร้าง DNA สาย leading
ก็จะเกิดการสังเคราะห์ดีเอ็นเอในอีกสายหนึ่ง แต่มีทิศทางตรงกันข้ามกับการเคลื่อนที่ของ
เรพลิเคชันฟอร์ค สำหรับการสังเคราะห์ดีเอ็นเอสายนี้จะสร้างได้เป็นสายสั้นๆ เนื่องจาก
เมื่อสร้างไปได้ระยะหนึ่งก็จะไปชนกับสายดีเอ็นเอที่สร้างไว้ก่อนหน้านี้ เมื่อจะเริ่มต้น
การสังเคราะห์ดีเอ็นเอสายสั้นๆ ในลักษณะนี้อีกก็ต้องรอให้เรพลิเคชันฟอร์คเคลื่อนห่าง
ออกไปก่อนสักระยะหนึ่ง จึงจะมีการเริ่มสร้างไพรเมอร์และเริ่มการสังเคราะห์ดีเอ็นเอสายสั้นๆ
นั้นอีก เรียกดีเอ็นเอสายสั้นๆ แต่ละสายนี้ว่าชิ้นส่วนโอกาซากิ (okazaki fragment)
และเรียกเรียกดีเอ็นเอสายสั้นๆ ที่วางเรียงรายกันในลักษณะเป็นสายยาวนี้ว่า สายตามหลัง
(lagging strand)

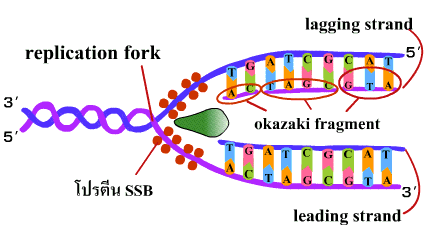
รูปที่ 1.13 การสร้าง DNA สาย lagging
ไพรเมอร์ออกโดยอาศัยคุณสมบัติของ 5'-3' เอ็กโซนิวคลีเอส (5'-3' exonuclease)
การกำจัดอาร์เอ็นเอไพรเมอร์ออกทำให้ดีเอ็นเอบริเวณดังกล่าวอยู่ในสภาพสายเดี่ยวที่ไม่มีคู่
อย่างไรก็ตามเอ็นไซม์ดีเอ็นเอโพลีเมอเรสเดิมจะนำดีออกซีไรโบนิวคลีโอไทด์ตัวใหม่เข้ามา
เติมเต็มช่องว่างนี้ ซึ่งการทำงานนี้อาศัยคุณสมบัติ 5'-3' โพลีเมอเรสและตามด้วยการเชื่อม
พันธะฟอสโฟไดเอสเทอร์ของชิ้นส่วนโอกาซากิ แต่ละโมเลกุลเข้าด้วยกันด้วยเอนไซม์ไลเกส
(ligase) ในที่สุดได้เป็น DNA ใหม่ 2 โมเลกุล โดยแต่ละโมเลกุลมีสายเดิมอยู่ 1 สาย

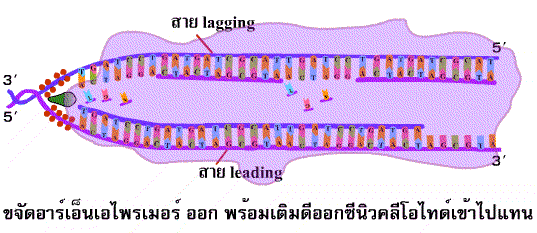

รูปที่ 1.14 การเชื่อม DNA สาย lagging
(สาย leading สร้างยาวขึ้นในทิศทางการขยายของ ทางแยกของการลอกแบบ ส่วนสาย
lagging สร้างยาวขึ้นไปทางด้านจุดเริ่มต้นของการสร้าง ซึ่งตรงข้ามกับการขยาย
ของทางแยกของการลอกแบบ) เชื่อว่าการสร้างสายใหม่ทั้ง 2 สายนี้เกิดขึ้นพร้อมกัน
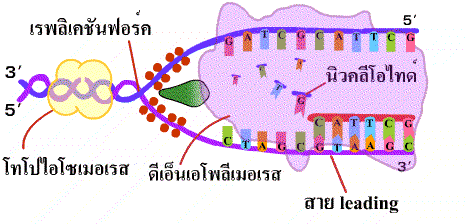
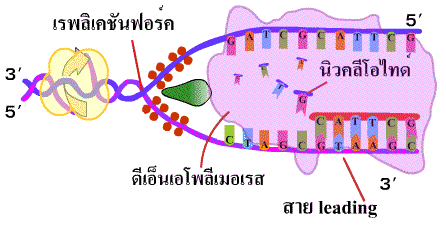
รูปที่ 1.15 การทำงานของเอ็นไซม์ดีเอนเอโพลีเมอเรส
ในการสร้างสาย lagging และ leading
ทั้งสองสายเหนือทางแยกของการลอกแบบเพื่อให้สามารถคลายเกลียวที่แน่นออกได้และ
เชื่อมต่อสายดีเอ็นเอกลับคืนสภาพเดิม
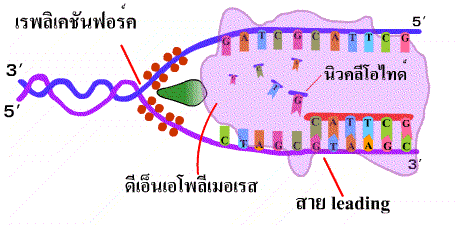
รูปที่ 1.16 การทำงานของเอ็นไซม์โทโปไอโซเมอเรส
จะเป็นแบบ 2 ทิศทางพร้อมกัน (bidirectional DNA replication) ทั้งในโปรคาริโอตและ
ยูคาริโอต

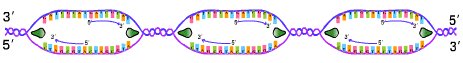
รูปที่ 1.17 การลอกแบบ DNA โดยเรพลิเคชันฟอร์ค เกิด 2 ทิศทางพร้อมกัน
(บน) ในโปรคาริโอตเกิดทีละตำแหน่งในโครโมโซมวงแหวน
(ล่าง) ในยูคาริโอตเกิดพร้อมกันหลายตำแหน่งในโครโมโซมแท่ง

